Series con raíz unitaria y procesos ARIMA
Contents
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import statsmodels.formula.api as smf
import seaborn as sns
from scipy.stats import t as t_Student
from joblib import Parallel, delayed
plt.style.use('seaborn')
4.3. Series con raíz unitaria y procesos ARIMA#
Series con raíz unitaria#
La caminata aleatoria (con o sin deriva) puede verse como un modelo AR(1) en el cual la raíz del polinomio de rezagos es uno:
Por ello, en general decimos que un proceso con tendencia estocástica tiene raíz unitaria.
Consideremos el siguiente proceso AR(2)
Su polinomio de rezagos es \(1-0.6\Lag - 0.4\Lag^2 = (1-0.4\Lag)(1-1\Lag)\), cuyas raíces son 1 y 2.5. Es decir, este proceso también tiene una raíz unitaria, y por tanto no es estacionario.
La factorización del polinomio de rezagos nos sugiere escribir
Visto como un proceso para \(y^*\equiv\Delta y_t\), este es un proceso AR(1) estacionario.
Procesos ARIMA(p,d,q)#
En el ejemplo anterior, como la primera diferencia de \(y_t\) es un proceso AR(1) estacionario, decimos que \(y_t\) es un proceso ARIMA(1,1,0).
En general, si un proceso integrado de orden \(d\), \(y_t\), es diferenciado \(d\) veces y su resultado \(\Delta^d y_t\) es un proceso ARMA(p,q), entonces decimos que \(y_t\) tiene un proceso ARIMA(p,d,q):
{admonition} Autorregresivo integrado de media móvil: ARIMA Sea \(\left\{\epsilon_t\right\}\) ruido blanco; el proceso estocástico \vspace{-0.5em}
\vspace{-0.5em} es llamado proceso ARIMA(p,d,q), donde \(\Phi(\Lag)\) es un polinomio de grado \(p\) cuyas raíces están fuera del círculo unitario, y \(\Theta(\Lag)\) es un polinomio de grado \(q\).
Repaso del modelo clásico de regresión lineal#
En el modelo clásico de regresión lineal se tiene
donde \(\epsilon_i\sim N(0,\sigma^2)\) es un error homoscedástico y no-autocorrelacionado.
Si se cumplen los supuestos del MCRL, este modelo se puede estimar de manera insesgada y eficiente por medio del estimador mínimos cuadrados ordinarios.
Para hacer un test sobre un parámetro
utilizamos el valor estimado por mínimos cuadrados ordinarios \(\hat{\beta}_j\) y su error estándar \(s.e.(\beta_j)\), y decimos que si la hipótesis nula es cierta entonces el estadístico
(tiene una distribución \(t\)-Student con \(n-k\) grados de libertad, donde \(n\) es el número de observaciones y \(k\) el número de parámetros estimados).
Es decir, con un nivel de significancia \(\alpha\), el intervalo
contendrá al valor estimado \(\hat{\beta}_j\) en \(100(1-\alpha)\%\) de las muestras siempre y cuando sea cierto que \(\beta_j=q\).
Por ello, cuando encontramos un valor \(\hat{\beta}_j\) que no está contenido en ese intervalo, rechazamos la hipótesis nula, y decimos que \(\beta_j\) es significativamente distinto de \(q\), a sabiendas de que nuestro procedimiento incurrirá en el error tipo-1 (rechazar una hipotésis verdadera) en \(100\alpha\%\) de las muestras.
Para que este procedimiento tenga validez, es necesario que el estadístico efectivamente tenga la distribución \(t\)-Student, lo cual es cierto siempre que se cumplan los supuestos del modelo clásico de regresión lineal.
Regresión espuria#
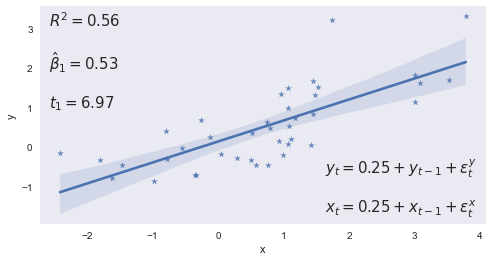
En 1974, Granger y Newbold demostraron, via simulaciones, que si una serie I(1) se estima en función de otra serie I(1) completamente independiente de ella, los estadísticos usuales tenderán a mostar que las dos series están relacionadas.
Es decir, si \(y_t, x_t\) son dos series I(1) independientes, al correr la regresión
fallaríamos en rechazar la hipótesis nula \(\beta_1 = 0\) (la cual es cierta porque \(y_t\) no depende de \(x_t\)) con una frecuencia mayor a la que sugiere la distribución \(t\)-student correspondiente:
Este fenómeno de encontrar relaciones inexistentes entre variables integradas se conoce como regresión espuria.
np.random.seed(240)
e = np.random.randn(40,2)
df = pd.DataFrame(
(0.25+e).cumsum(axis=1),
columns=['y', 'x']
)
res = smf.ols("y ~ x" , df).fit()
fig, ax = plt.subplots(figsize=[8,4])
sns.regplot(x='x',y='y',data=df, marker='*',ax=ax)
ax.set(xlabel='x', ylabel='y')
statslabel = f'$R^2={res.rsquared:.2f}$'
statslabel += f'\n\n$\\hat\\beta_1={res.params[1]:.2f}$'
statslabel += f'\n\n$t_1={res.tvalues[1]:.2f}$'
ax.annotate(statslabel, (0.02,0.98), xycoords='axes fraction', size=15, va='top')
modellabel = r'$y_t = 0.25 + y_{t-1} + \epsilon^y_t$'
modellabel += '\n\n$x_t = 0.25 + x_{t-1} + \\epsilon^x_t$'
ax.annotate(modellabel, (0.98,0.02), xycoords='axes fraction', size=15, va='bottom',ha='right')
ax.grid(False);
res.summary()
| Dep. Variable: | y | R-squared: | 0.561 |
|---|---|---|---|
| Model: | OLS | Adj. R-squared: | 0.550 |
| Method: | Least Squares | F-statistic: | 48.60 |
| Date: | Thu, 21 Jul 2022 | Prob (F-statistic): | 2.69e-08 |
| Time: | 00:15:57 | Log-Likelihood: | -40.091 |
| No. Observations: | 40 | AIC: | 84.18 |
| Df Residuals: | 38 | BIC: | 87.56 |
| Df Model: | 1 | ||
| Covariance Type: | nonrobust |
| coef | std err | t | P>|t| | [0.025 | 0.975] | |
|---|---|---|---|---|---|---|
| Intercept | 0.1400 | 0.119 | 1.174 | 0.248 | -0.101 | 0.381 |
| x | 0.5325 | 0.076 | 6.972 | 0.000 | 0.378 | 0.687 |
| Omnibus: | 7.474 | Durbin-Watson: | 2.329 |
|---|---|---|---|
| Prob(Omnibus): | 0.024 | Jarque-Bera (JB): | 6.173 |
| Skew: | 0.843 | Prob(JB): | 0.0457 |
| Kurtosis: | 3.929 | Cond. No. | 1.94 |
Notes:
[1] Standard Errors assume that the covariance matrix of the errors is correctly specified.
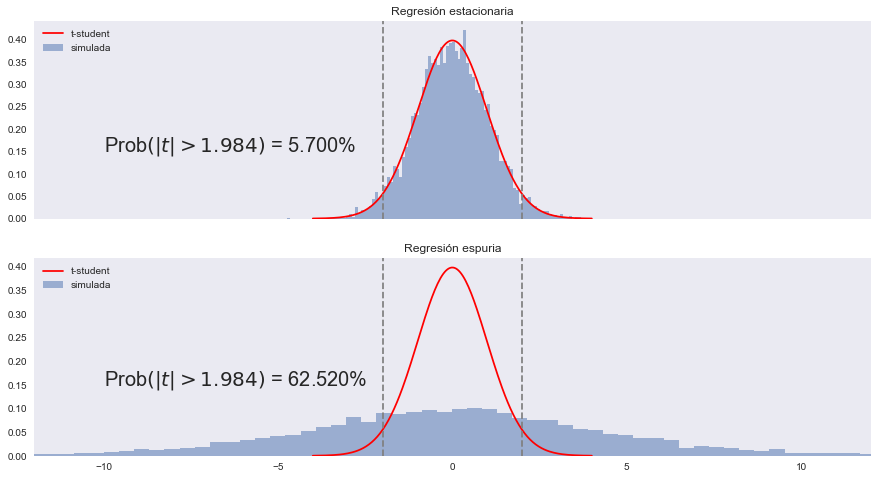
Experimento de Monte Carlo para determinar la distribución del estadítico t
%%time
def regresion(T=40):
e = np.random.randn(2, T)
df = pd.DataFrame(dict(
x = e[0].cumsum(),
y = e[1].cumsum(),
u = e[0],
v = e[1]
),
columns=['x', 'y', 'u', 'v']
)
t_estacionario = smf.ols("v~u", data=df).fit().tvalues[1]
t_espurio = smf.ols("y~x", data=df).fit().tvalues[1]
return t_estacionario, t_espurio
def Monte_Carlo(funcion, repeticiones, columns, *args, **kwargs):
datos = Parallel(n_jobs=-1)(delayed(funcion)(*args, **kwargs) for _ in range(repeticiones))
return pd.DataFrame(datos, columns=columns)
N = 10_000
np.random.seed(12345)
tvalues = Monte_Carlo(regresion, N, ['estacionaria', 'espuria'])
maxtval = 12
t5crit = t_Student.isf(0.05/2, 98)
fig, (ax0,ax1) = plt.subplots(2,1,figsize=[15, 8], sharex=True)
def plot_simulated(caso, ax):
ff, edges = np.histogram(tvalues[caso], bins=100, density=True)
center = (edges[1:] + edges[:-1])/2
w = center[1] - center[0]
ax.bar(center, ff, width=w, alpha=0.5)
xvals = np.linspace(-4,4, 200)
ax.plot(xvals, t_Student.pdf(xvals, df=98), '-r')
ax.set(xlim=[-maxtval, maxtval], title=f'Regresión {caso}')
ax.legend(['t-student', 'simulada'], loc='upper left')
# valores críticos
opciones = dict(ls="--", color='gray')
ax.axvline(t5crit, **opciones)
ax.axvline(-t5crit, **opciones)
# error tipo I
err1 = (tvalues[caso].abs() > t5crit).mean()*100
ax.annotate(f'Prob$\\left(|t| > 1.984\\right)$ = {err1:.3f}%', (-10,0.15), size=20)
ax.grid(False)
plot_simulated('estacionaria', ax0)
plot_simulated('espuria', ax1)
CPU times: total: 51.7 s
Wall time: 2min 7s
Regresión lineal con series integradas#
¿Cómo estudiar la relación entre series integradas sin incurrir en regresiones espurias?
Supongamos que tanto \(y_t\) como \(x_t\) son series I(1).
Entonces, por definición, \(\Delta y_t\) y \(\Delta x_t\) son series estacionarias.
En tal caso, el modelo
puede estimarse por mínimos cuadrados, y la prueba \(\beta_1=0\) se realiza usando el procedimiento usual.
Una alternativa mejor, que estudiaremos con detalladamente más tarde en el curso, es estimar la regresión en niveles
y determinar si los residuos de esta regresión son estacionarios. Si lo son, diremos que \(y_t\) está cointegrada con \(x_t\).
Procesos AR(p) con raíz unitaria#
Recordemos que el proceso AR(p) puede escribirse
Supongamos que los coeficientes autorregresivos suman uno. Entonces
Es decir, si \(\phi_1 + \phi_2 + \dots + \phi_p=1\), entonces el proceso tiene raíz unitaria.


